Qualität in der Hochdurchsatz-Genotypisierung ist machbar

© Zubada - stock.adobe.com
Dank der Entwicklung von Hochdurchsatzverfahren zur Genotypisierung stehen aus den Genomforschungsprojekten der vergangenen Jahre unvorstellbare Datenmengen zur Verfügung. "Ich bin sicher, dass bisher erst ein Bruchteil der wissenschaftlichen Fragestellungen, für die diese Daten generiert wurden, beantwortet wurde. Ein Großteil der beantwortbaren Fragen ist bis heute vermutlich noch nicht einmal gestellt worden." Diese Einschätzung formulierte Prof. Dr. Michael Krawczak (Universität Kiel) im Rahmen der Informationsveranstaltung zum Qualitätsmanagement von Genotypisierungsdaten, die am 21. Juni 2010 in Berlin stattfand. In der Veranstaltung, an der rund 50 Molekulargenetiker und Bio-Informatiker aus ganz Deutschland teilnahmen, wurden die Ergebnisse eines Projektes vorgestellt, das vom Bundesministerium für Bildung und Forschung (BMBF) von 2008 bis 2010 im Rahmen der Methodenausschreibung gefördert worden war.
Fragen der Qualitätssicherung von Daten, die bei der Hochdurchsatz-Genotypisierung generiert werden, reichen von Problemen der Validität und Plausibilität über die Erkennung und Vermeidung von Fehlern bis hin zu Anforderungen an Datenhaltung und Datentransfer. Gemäß dem Motto der TMF - "das Rad nicht immer wieder neu erfinden" - hat die Projektgruppe die verschiedensten Verfahren der Verarbeitung und Analyse von Genotypisierungsdaten systematisch nach Qualitätsgesichtspunkten geprüft, verglichen und bewertet.
Präsentiert wurden einerseits Empfehlungen zur Wahl geeigneter Qualitätssicherungsverfahren, andererseits aber auch konkrete Hilfsmittel in Form von Begleitdokumenten oder Software-Tools. In der Veranstaltung wurden wesentliche Aussagen und Ergebnisse aus den einzelnen Teilprojekten hervorgehoben:
- Calling-Algorithmen: Insgesamt wird CRLMM als der beste Allel-Calling-Algorithmus empfohlen. JAPL kommt in Frage, wenn möglichst viele Personen in den Analysen berücksichtigt werden müssen, während Chiamo die meisten Vorteile bietet, wenn viele SNPs in den Analysen verwendet werden sollen.
- Es wurde gezeigt, dass sich die Analyse von Cluster-Plots anhand der drei Kriterien „Kompaktheit“, „Verbundenheit“ und „Trennbarkeit“ erfolgreich algorithmisieren lässt. Die Ergebnisse werden als R-Paket verfügbar gemacht.
- SNP-Daten müssen vor einer Weitergabe an Dritte oder zur kooperativen Bearbeitung mit Begleitinformationen – beispielsweise zum DNA-Sample, zum Genotypisierungsprozess oder zur Patienteneinwilligung – versehen werden. Zur Erfassung solcher Daten wurde ein konsentiertes Formular erstellt.
- Bei Software für die Aufbereitung von SNP-Daten konnten deutliche (Geschwindigkeits-)Vorteile für das Programm GenABEL nachgewiesen werden.
- Eine konsentierte Nomenklatur für nicht-kanonische Intensitäts-Cluster von SNPs auf Illumina Genotypisierungschips wurde erstellt. Sie dient als Grundlage für eine Verbesserung automatisierter Calling-Verfahren, um nicht nur drei sondern auch vier, fünf und mehr Cluster korrekt zu erfassen und Genotypen mit höherer Präzision zu erhalten.
- Es wurde gezeigt, dass Scores zur Clustervalidierung geeignet sind, um fehlerhafte Genotypen zu identifizieren. Kriterien wie Call Rate, MAF oder HWE sind unverzichtbar und komplementär zu Scores. Der „silhouette score“ wird als unabhängiges Kriterium weiter empfohlen.
- Für eine standardisierte und systematische Qualitätskontrolle von Replikations-Genotypisierungsdaten wurde im Rahmen des Projektes die Software „RepliCheckSNP“ entwickelt und in der Veranstaltung vorgeführt.
- Zur Speicherung von Genotypisierungsdaten werden de-normalisierte BLOBs („Binary Large Objects“) empfohlen.
- Datenschutzempfehlungen beinhalten unter anderem die Einbindung von Datentreuhändern.
Als Genotypisierung bezeichnet man die Analyse des Genotyps, also der molekular kodierten Erbanlagen eines Organismus. Sie ist die entscheidende molekulargenetische Methode im Rahmen so genannter Assoziationsstudien, die zur Identifizierung von „Krankheitsgenen“ dienen. Dabei werden die Erbanlagen einer möglichst großen Gruppe von Personen, die eine bestimmte Krankheit ausgebildet haben, mit den Erbanlagen einer nicht von der Krankheit betroffenen Gruppe verglichen. Auf Basis einer DNA-basierten Genotypisierung lassen sich beispielsweise Arzneimittel gezielt für bestimmte Patientengruppen einer spezifischen genetischen Ausprägung entwickeln.
Impressionen

Die Referenten der Informationsveranstaltung am 21. Juni 2010. V.l.n.r.: Dr. Thomas Bettecken, Prof. Dr. Thomas Wienker, Prof. Dr. Andreas Ziegler, Dr. Arne Pfeufer, Prof. Dr. Michael Krawczak, Dr. Andresas Wolf, Prof. Dr. Bertram Müller-Myhsok, Dr. Michael Steffens, Mathias Freudigmann. © TMF e.V.

Mathias Freudigmann (Geschäftsstelle TMF e.V.), Leiter des Teilprojektes Koordination, sprach einleitende Worte und dankte dem Förderer BMBF ebenso wie allen Projektmitwirkenden. © TMF e.V.

Prof. Dr. Michael Krawczak (Universität Kiel) führte ins Thema ein und berichtete über die Hintergründe und den Werdegang des Projekts. © TMF e.V.
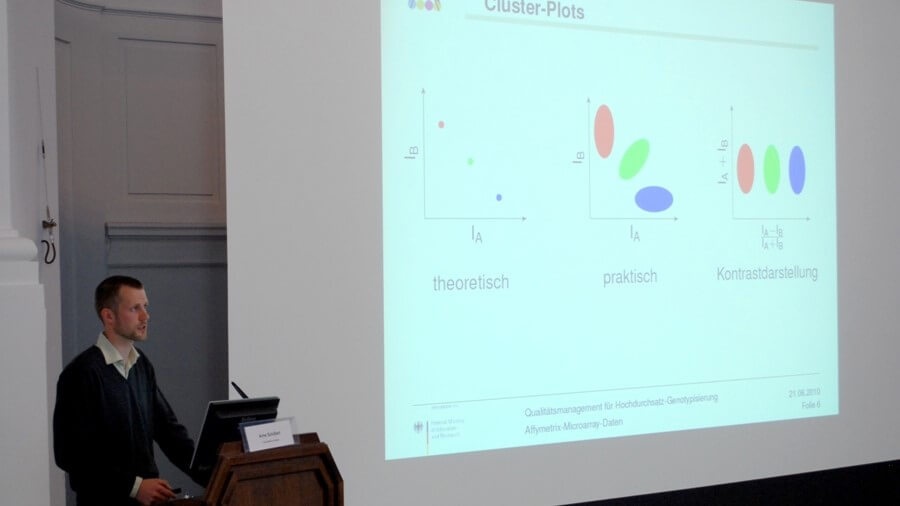
Dr. Arne Schillert (Universität Lübeck) stellte die Projektergebnisse zur automatischen Beurteilung von Cluster-Plots sowie zu Verfahren des Genotyp-Callings vor. © TMF e.V.
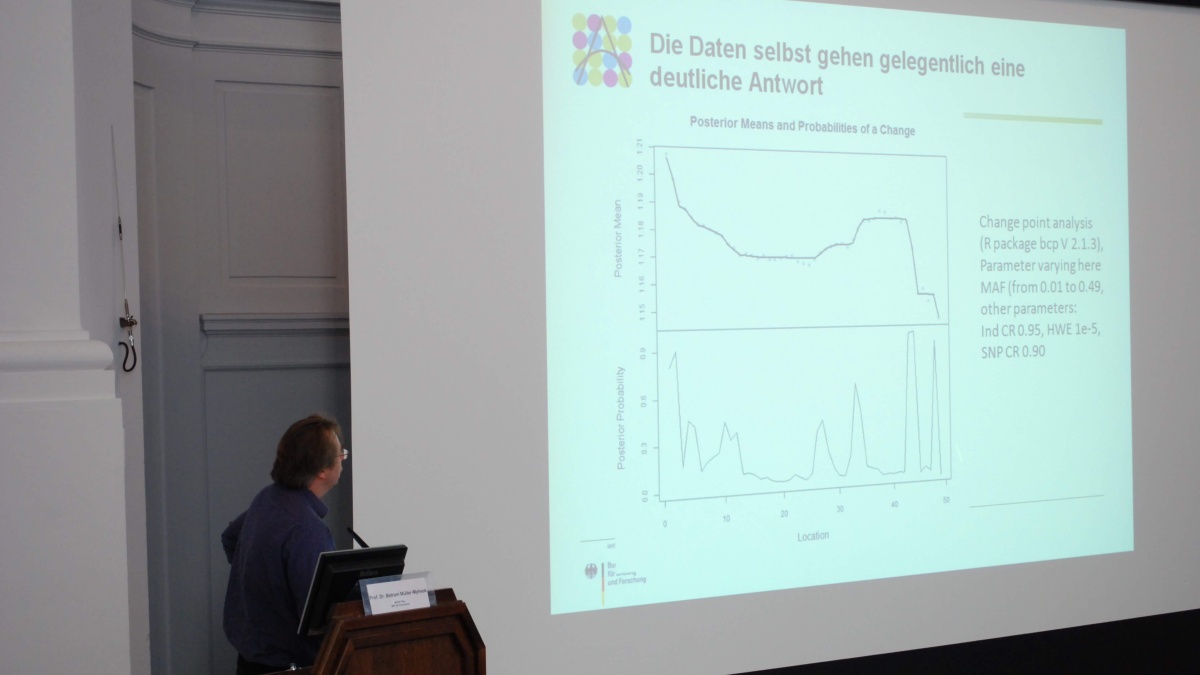
Prof. Dr. Betram Müller-Myhsok (MPI für Psychiatrie, München) © TMF e.V.

Prof. Dr. Betram Müller-Myhsok (MPI für Psychiatrie, München) ... © TMF e.V.

... und Dr. Thomas Bettecken bewerteten Kriterien zur Plausibilitätsprüfung. © TMF e.V.

Fehlererkennung und -korrektur beim Umgang mit Genotypisierungsdaten war das Thema von Dr. Michael Steffens ... © TMF e.V.
... und Prof. Dr. Thomas Wienker (Universität Bonn). © TMF e.V.

Andreas Wolf (Universität Kiel) erläuterte die Anforderungen an Datenhaltung und -transfer. © TMF e.V.

Mit 'RepliCheckSNP' steht nun ein Software-Werkzeug zur standardisierten und systematischen Qualitätskontrolle von Genotypisierungsdaten aus Replikationsstudien zur Verfügung. Dr. Arne Pfeufer führte das Programm vor. © TMF e.V.

Rund 50 Molekulargenetiker und Bio-Informatiker aus ganz Deutschland waren der Einladung zu der Informationsveranstaltung nach Berlin gefolgt. © TMF e.V.
Vortragsfolien zum Download
| Anhang | Size |
|---|---|
| Programmflyer "Qualitätsmanagement für Hochdurchsatz-Genotypisierung" | 1.29 MB |
| Anhang | Size |
|---|---|
| Prof. Dr. Michael Krawczak (Christian-Albrecht-Universität Kiel): Einführung | 373.6 KB |
| Anhang | Size |
|---|---|
| Dr. Arne Pfeufer (HMGU München): Daten- und Qualitätsmanagement von Replikationsdaten | 246.43 KB |
| Anhang | Size |
|---|---|
| Dr. Thomas Bettecken (Max-Planck-Institut): Plausibilitätskriterien | 773.51 KB |


